(Application) MPIを用いた分割ファイルの解析
作成者: 西澤誠也
作成日: 2014/03/13
複数プロセスで解析を行う
- メリット
- 処理が重たい場合、複数のCPUを用いることで早く解析を終わらせることが出来る
- Ruby のスレッドは、基本的には排他的に動作するため、複数のCPUを同時に使うことは出来ない
- プロセスあたりの同時利用ファイル数やCPU時間の制限があるシステムで、1プロセスで実行した場合にそれらの制限に引っかかる場合は、複数プロセスに分散させることで制限を回避することが出来る
- ストレージの構成によっては、同時に複数のファイルにアクセスすることでスループットを上げる事が出来る
- 処理が重たい場合、複数のCPUを用いることで早く解析を終わらせることが出来る
- デメリット
- MPI など、プロセス間のデータ交換が必要
MPI を用いた分割ファイルの解析
- 数値シミュレーションモデルによっては、分割ファイルを出力する物があり、それらの解析の簡単な例を示す。
分割ファイルを作成する
- 下のサンプルプログラム用のデータを作る
- gphys に同梱されているデータを元データとする: testdata/T.jan.nc
- Ruby スクリプト mksubset.rb
require "numru/gphys"
include NumRu
# open file
t = GPhys::IO.open("T.jan.nc","T")
nx, ny, nz = t.shape
# devide into 4 (=2x2) subsets
sub = Array.new(4)
sub[0] = t[0...nx/2, 0...ny/2, true]
sub[1] = t[nx/2..-1, 0...ny/2, true]
sub[2] = t[0...nx/2, ny/2..-1, true]
sub[3] = t[nx/2..-1, ny/2..-1, true]
# write files
4.times do |i|
fname = "T.jan.pe%06d.nc"%i
file = NetCDF.create(fname)
GPhys::IO.write(file, sub[i])
file.close
end
- 実行
% ruby mksubset.rb
- 結果
以下の4つのファイルが作られる
T.jan.pe000000.nc T.jan.pe000001.nc T.jan.pe000002.nc T.jan.pe000003.nc
MPIを用いて分割ファイルを解析する
- Ruby-MPI のインストールが必要
% sudo gem install ruby-mpi
- Ruby スクリプト anal_mpi.rb
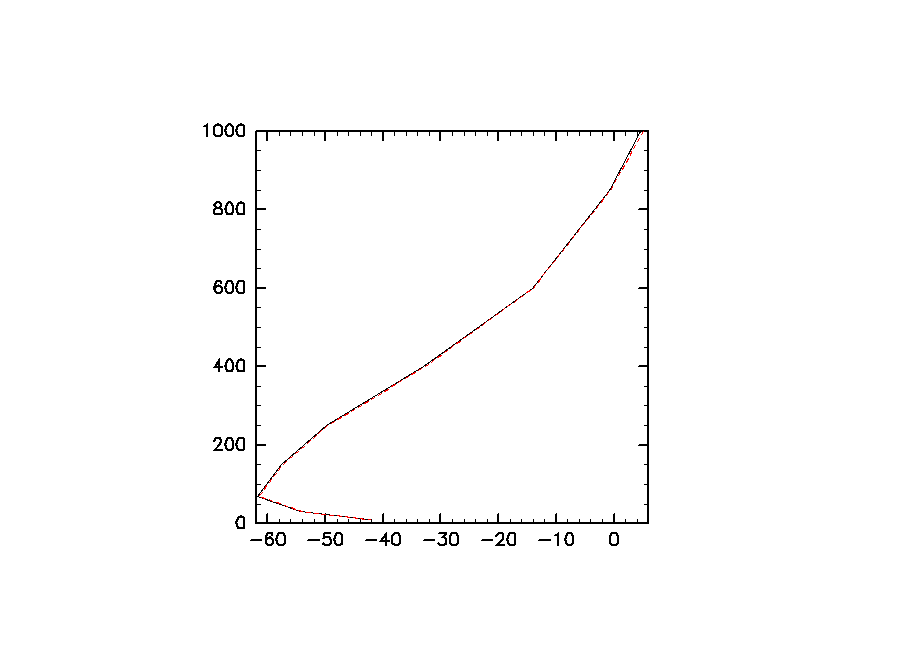
- 水平平均の鉛直プロファイルを作成する処理プログラムです。ファイル読み込みおよび水平平均を並列処理で行っています。
require "numru/gphys"
require "mpi"
include NumRu
NFILES = 4
MPI.Init
world = MPI::Comm::WORLD
rank = world.rank
size = world.size
nf = NFILES/size
fnames = Array.new
for i in nf*rank...[nf*(rank+1),NFILES].min
fnames.push "T.jan.pe%06d.nc"%i
end
puts "rank #{rank}: #{fnames.inspect}"
# open files
t = GPhys::IO.open(fnames, "T")
# horizontal mean of the subsets
tm = t.mean(0,1)
# get narray
tm = tm.val.get_array!
# buffer for recieved data
if rank == 0 # only rank 0 recieve data
buf = NArray.new(tm.typecode, tm.length)
else
buf = nil
end
# execute reduction (MPI communication)
world.Reduce(tm, buf, MPI::Op::SUM, 0)
# graph
if rank == 0 # only rank 0
buf /= size # sum to mean
require "numru/dcl"
z = t.coord("level").val
DCL.gropn(1)
DCL.grfrm
DCL.usgrph(buf, z) # black line
# for comparison with the original data
to = GPhys::IO.open("T.jan.nc", "T").mean(0,1).val.get_array!
DCL.sgplzu(to, z, 3, 21) # red broken line
DCL.grcls
end
MPI.Finalize
- 実行 (プロセス数は任意, ここでは例として4としている)
% mpirun -np 4 ruby anal_mpi.rb
- 結果
キーワード:
参照: